PyMOL是一款基于Python语言的三维结构可视化软件,非常简单实用。现将Pymol用于分析蛋白与蛋白相互作用区域教程总结下来。
1.安装PyMOL软件,有两种来源:一是官网版,二是开源版。
1.1 官网版安装
官网:https://pymol.org/2/ ,官网安装,方便快捷,但需要注册,并支付相应费用。或者可以选择试用30天。30天后,一般功能不受影响,但渲染(Ray)后会出现水印,无法生成高质量的图片。
1.2 开源版可以通过网络上相关教程下载安装。
2. 下载蛋白复合物PDB文件:这里以8K4U( BtKY72 spike receptor-binding domain (RBD) complexed with bat ACE2)为例子进行分享。
2.1 打开PDB官网https://www.rcsb.org/ ,搜索框输入8K4U,点击搜索,找对应PDB结构。
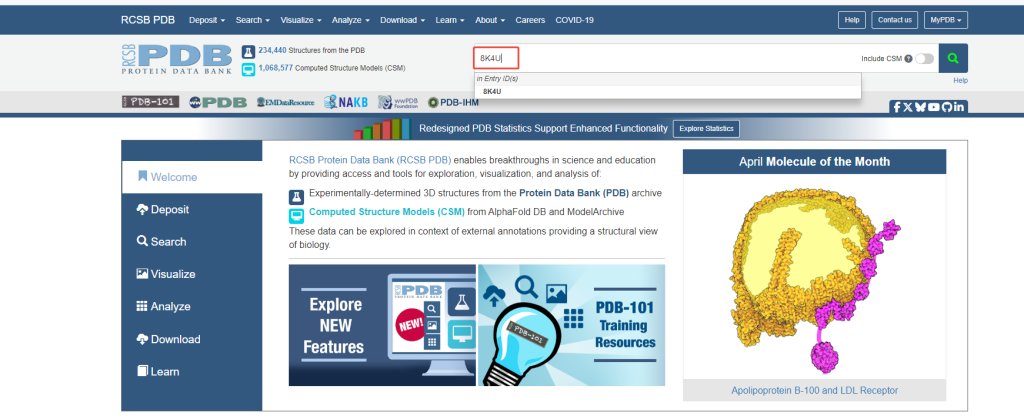
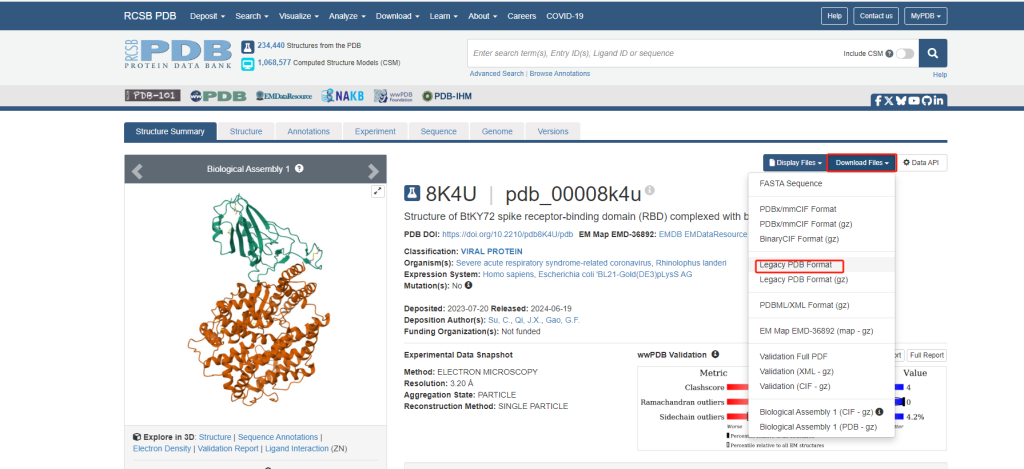
2.2 下载PDB格式文档,点击Download Files-Legacy PDB Format,下载文件保存在合适位置。
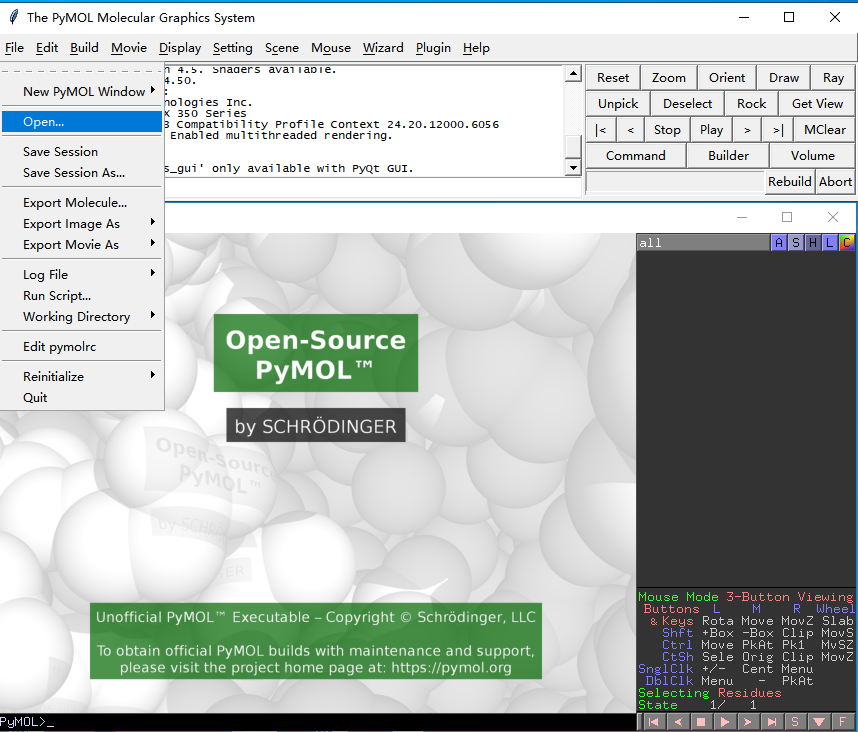
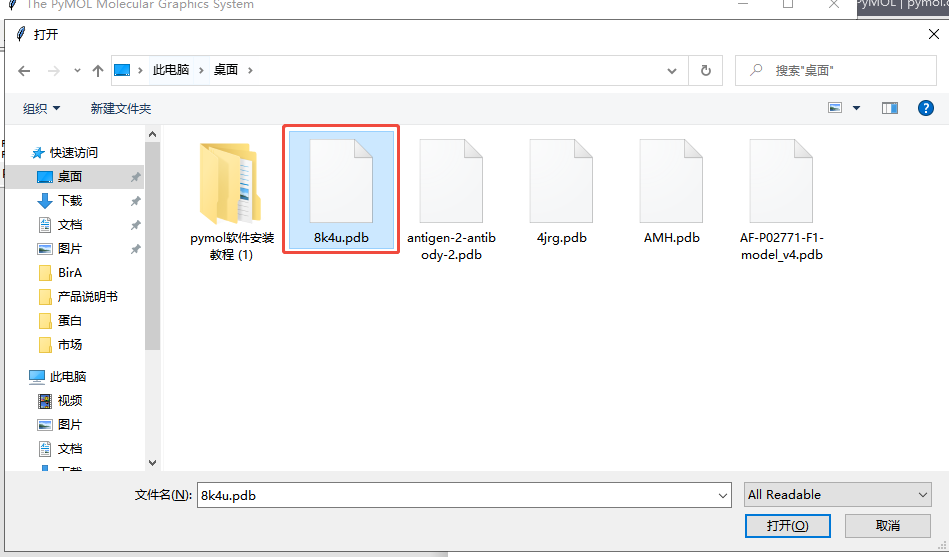
3. 导入PDB格式文件,打开PyMOL软件,点击File-Open-选择已下载的8K4U文件,3D图像可以通过鼠标拖动旋转。
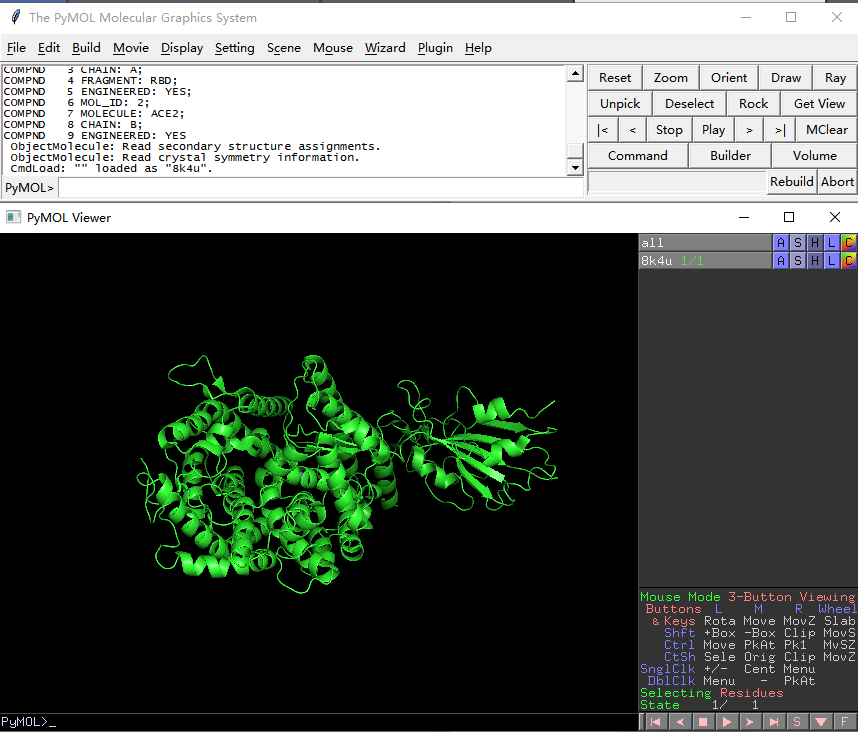
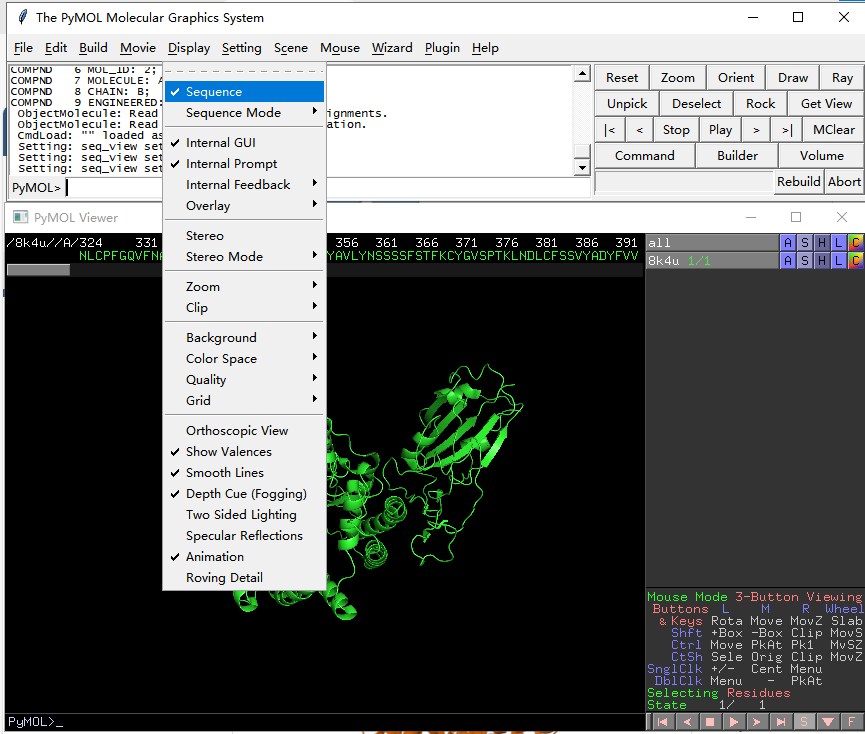
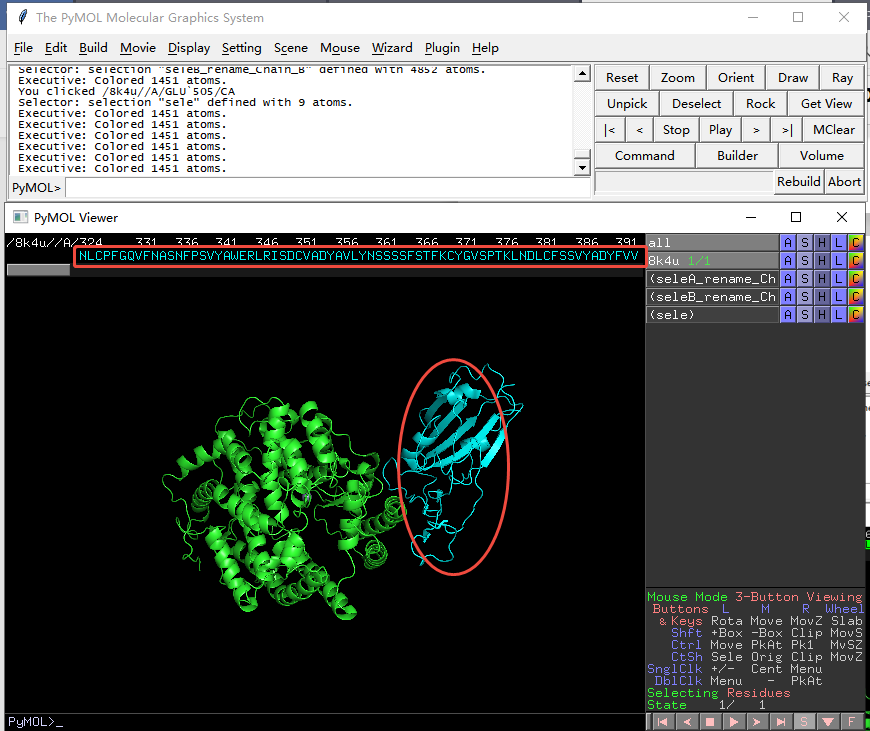
4. 序列查看:点击菜单栏Display-sequence,即可展示氨基酸序列;
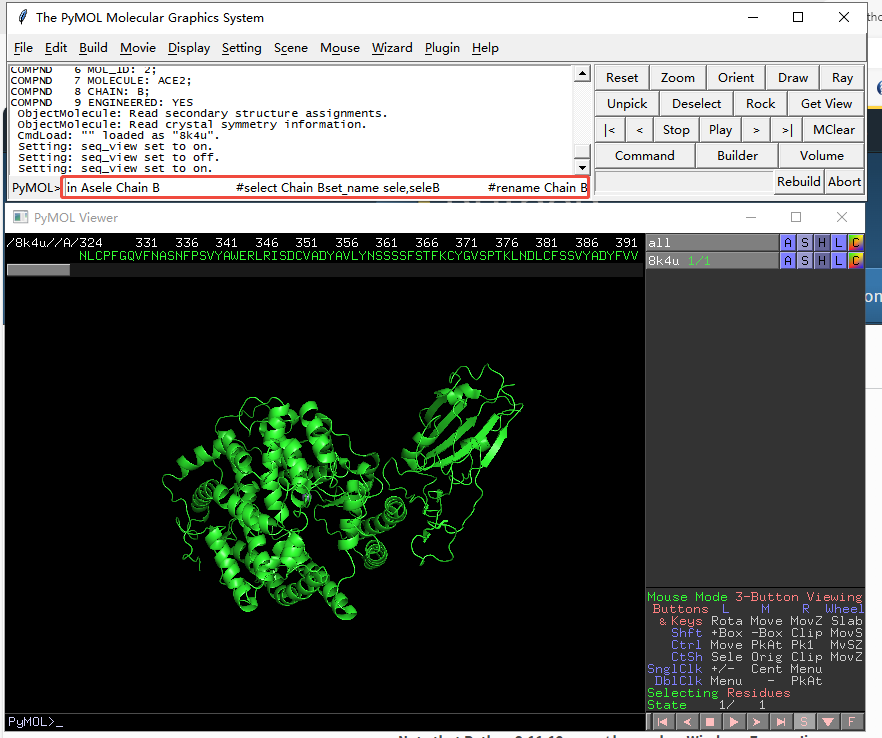
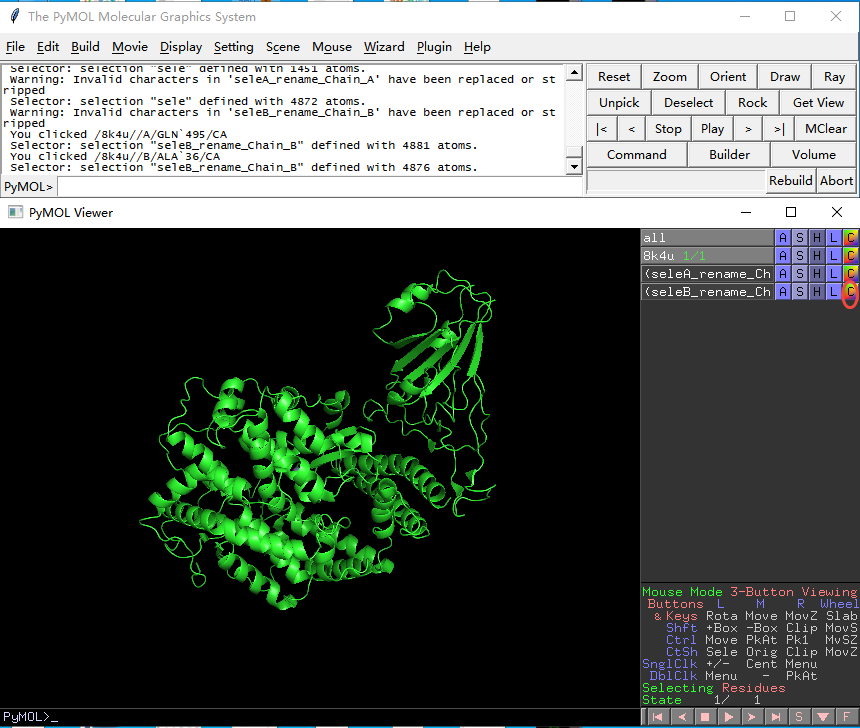
5. 设置显示两条链,在命令行键入如下命令(如果是两条蛋白序列,默认的名称是A和B),点击Enter键,右侧面板会增加seleA_rename_Chain A,seleB_rename_Chain B选项。
sele Chain A #select Chain A
set_name sele,seleA #rename Chain A
sele Chain B #select Chain B
set_name sele,seleB #rename Chain B
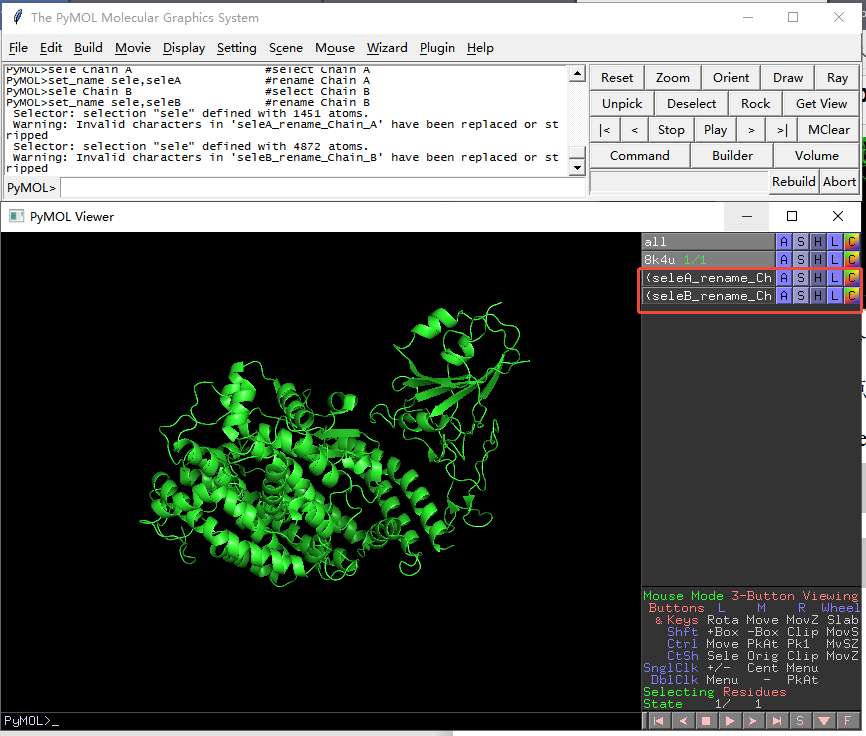
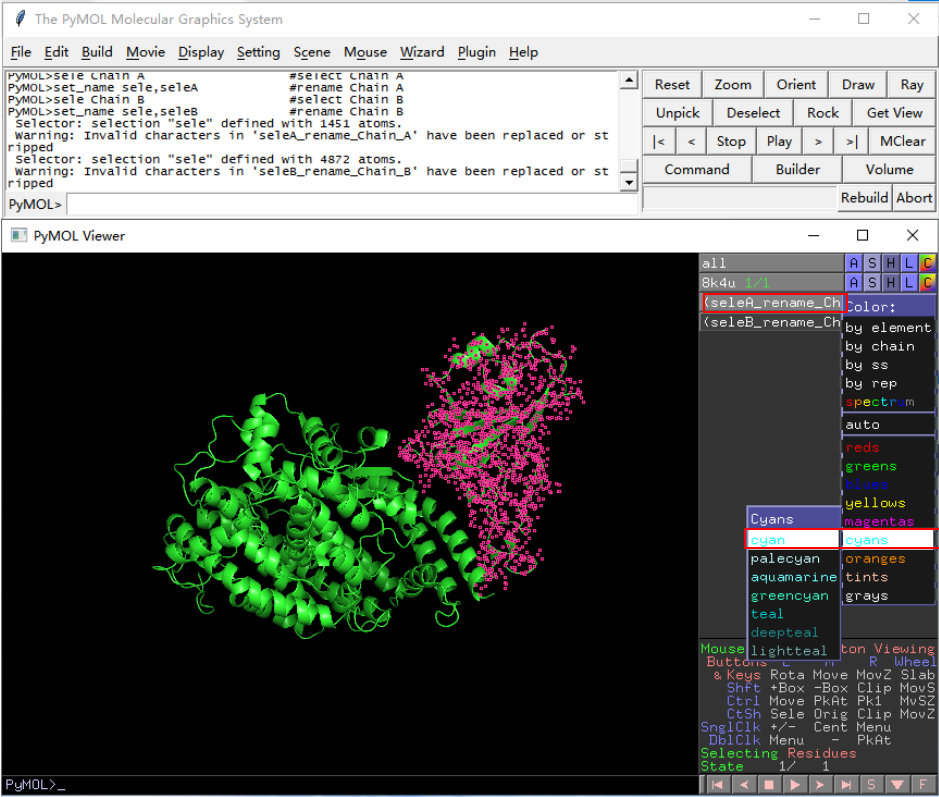
6. 设置两条链颜色,点击右侧面板seleA_rename_Chain A,点击“C”按钮,选择一种颜色,比如Cyan,选择后Chain A序列和3D结构图都变成Cyan颜色。
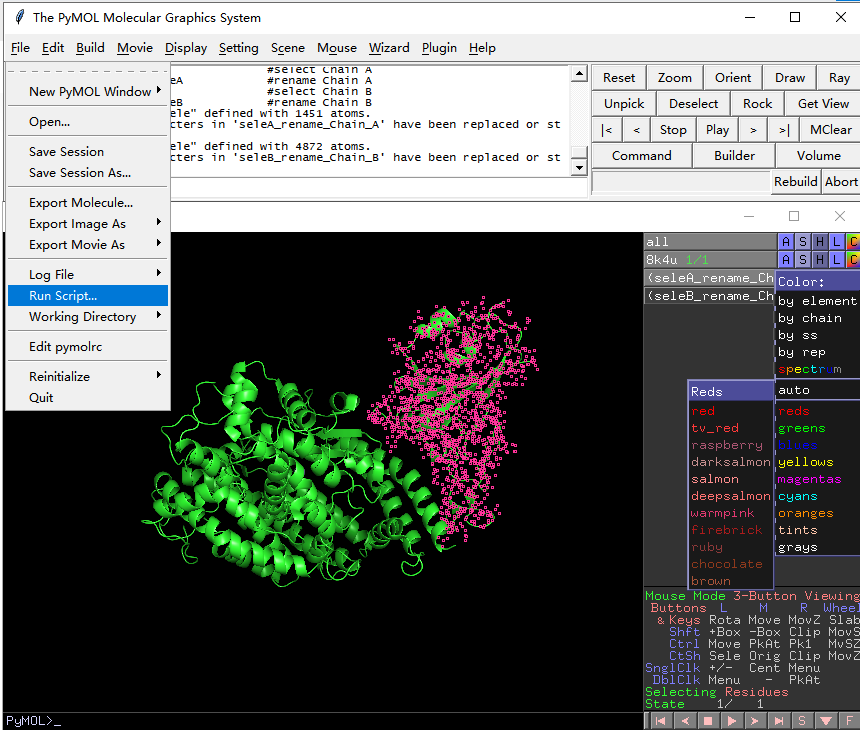
7.下载InterfaceResidues.py脚本保存到本地,命名为InterfaceResidues.py,点击菜单栏file-run script -InterfaceResidues.py, 运行脚本。
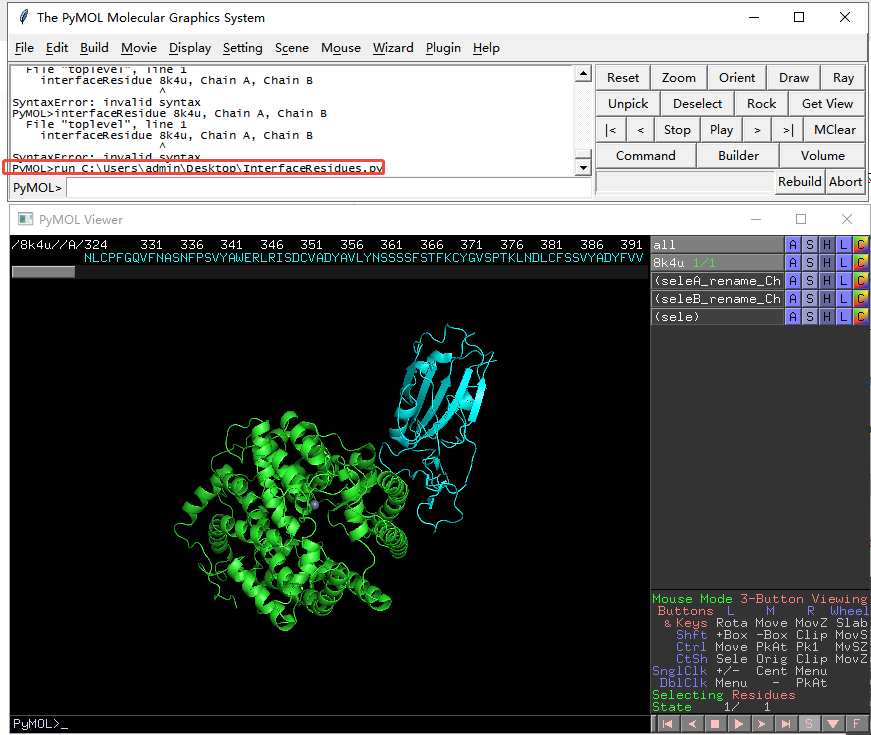
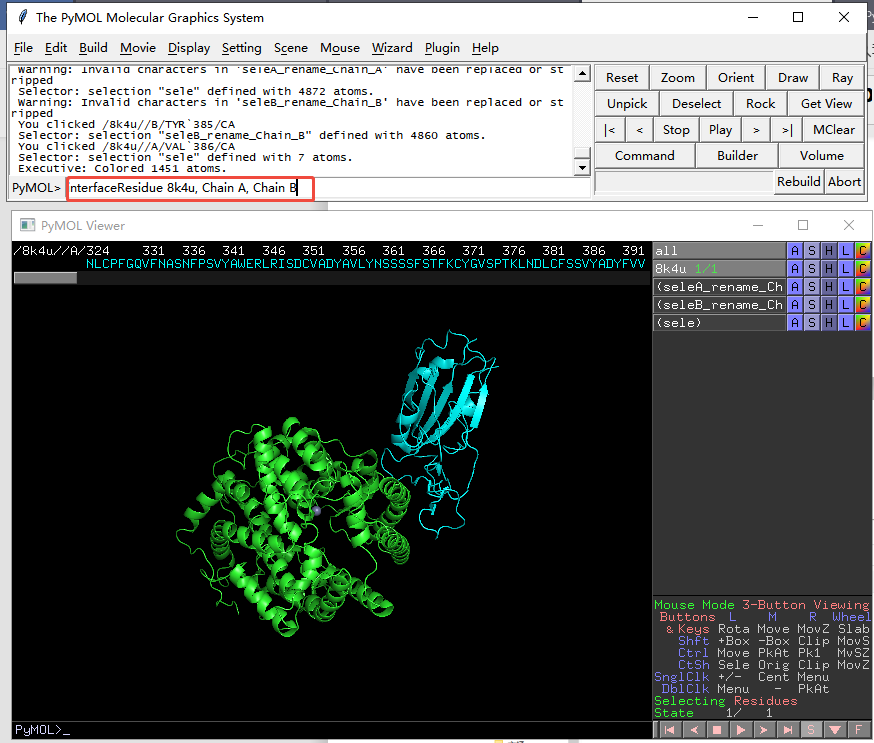
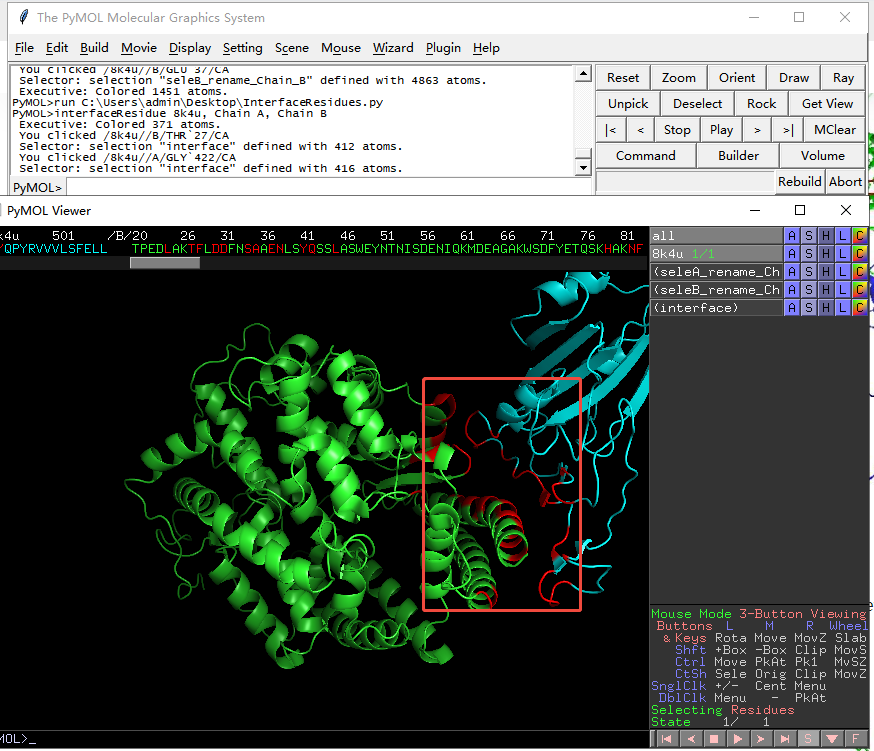
8. 查看蛋白互作区域:在命令行键入命令:interfaceResidue 8k4u, Chain A, Chain B (8k4u替换成你的projectname,即PDB文档名),然后点击Enter键,右侧面板出现interface选项。
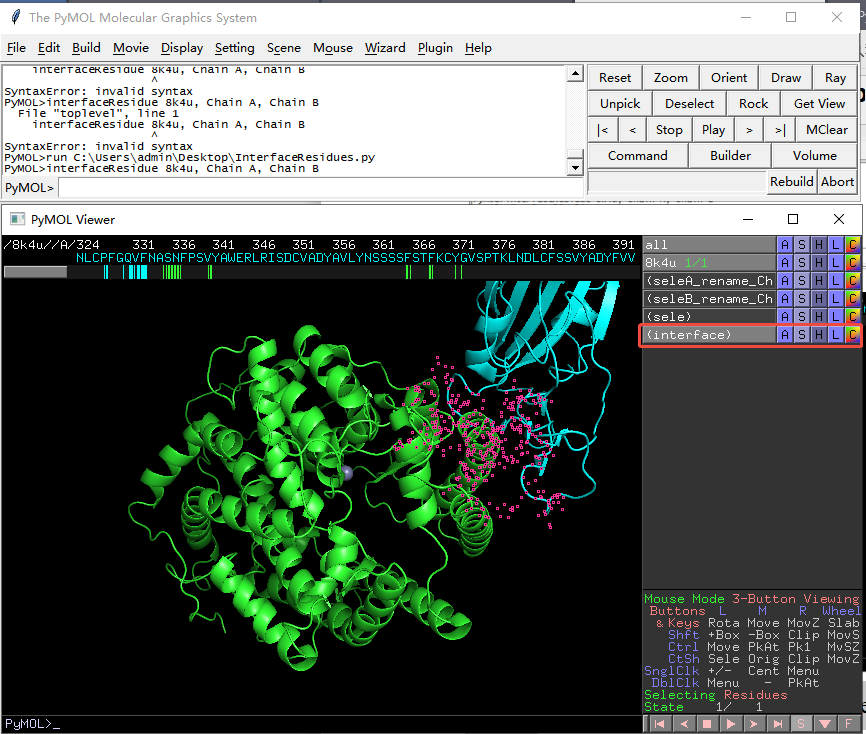
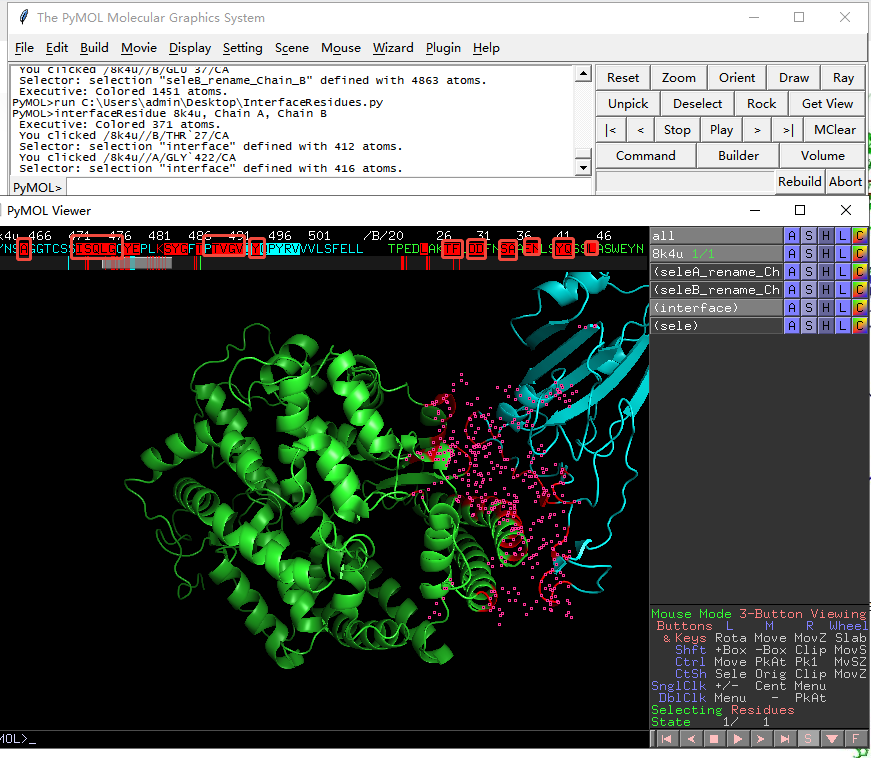
9. 点击interface选项,再点击“C”按钮,选择颜色,这里选择Red,3D图像的红色位置即为互作区域,在sequence panel可以看到具体哪些氨基酸互作(红色的为互作氨基酸)。
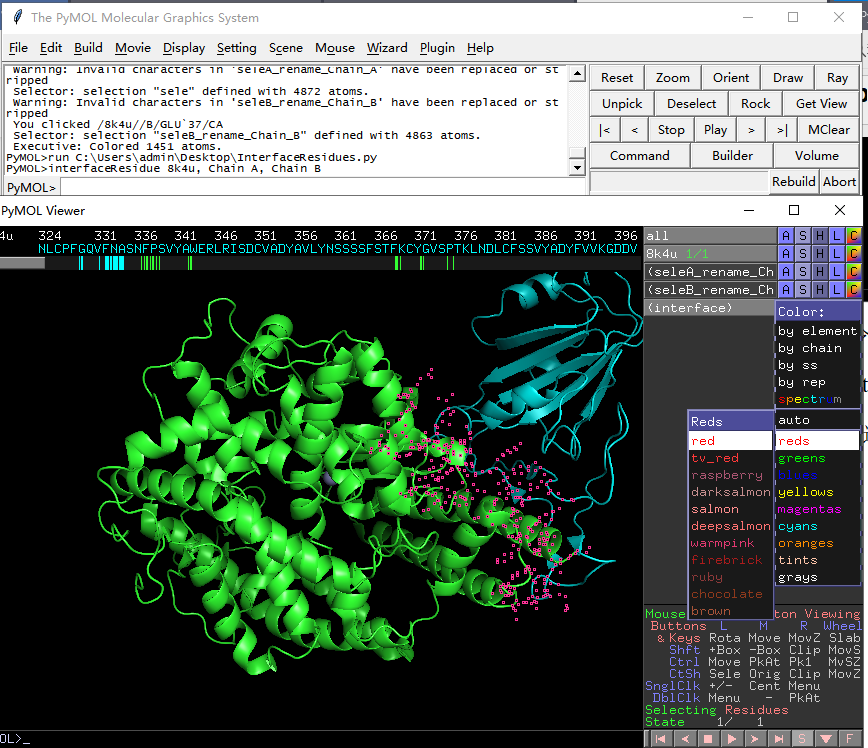
希望这篇科普文能帮助你快速学会利用PyMOL分析蛋白互作及互作区域。

