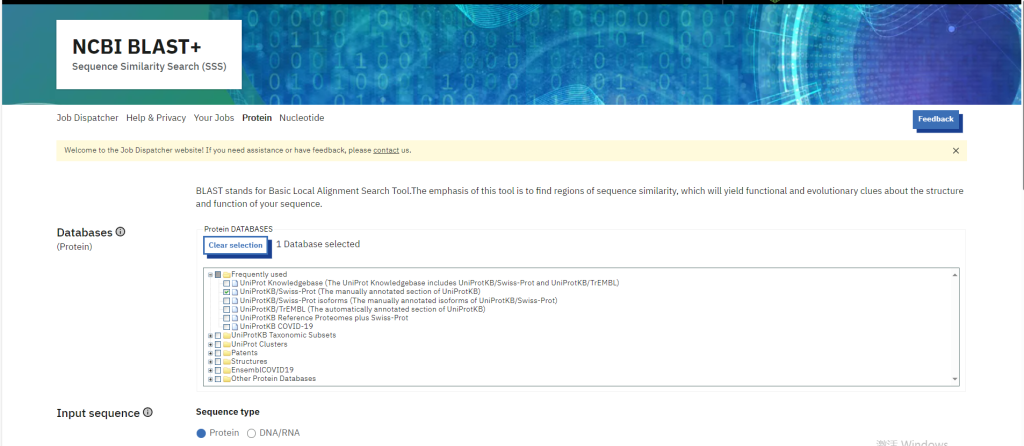
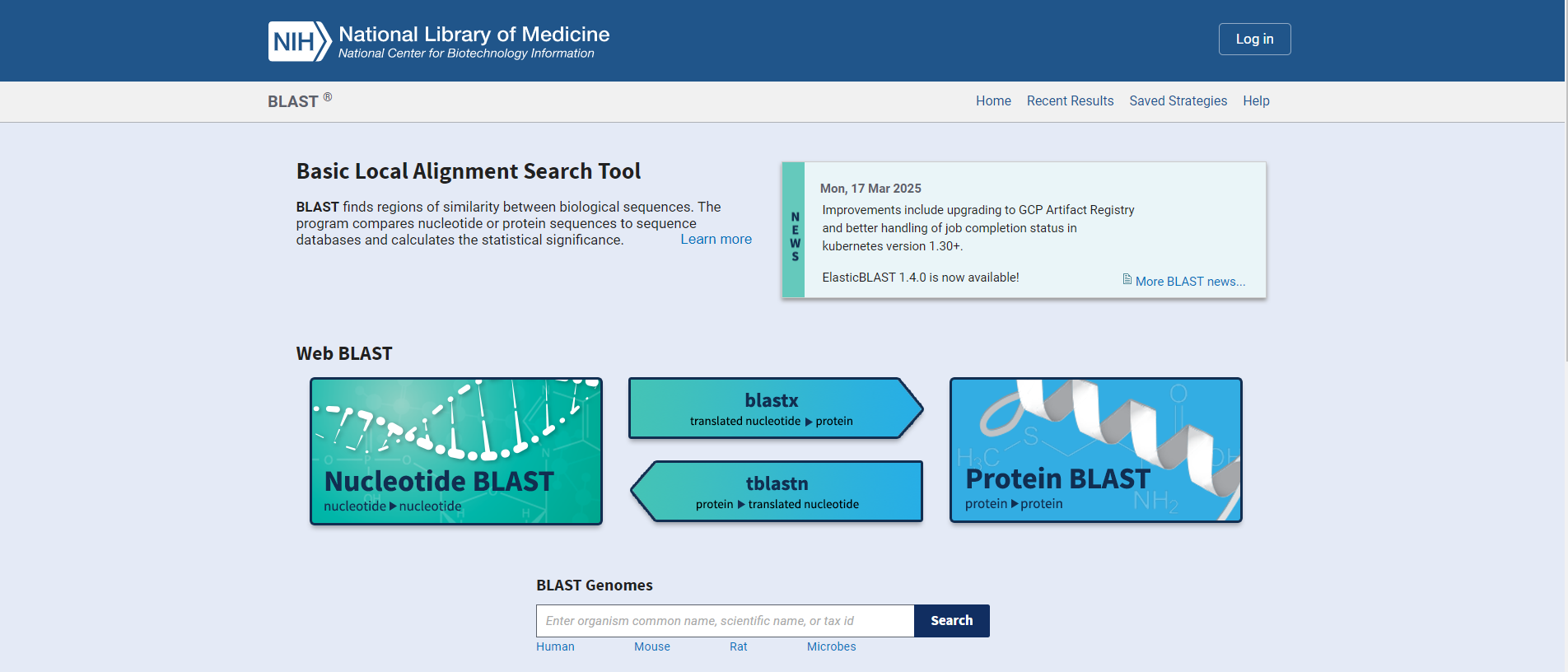
在生物信息学研究中,序列比对是基础且关键的分析步骤。以下是常用的氨基酸序列比对在线工具和数据库。
1. NCBI Protein BLAST (blastp)
网址: https://blast.ncbi.nlm.nih.gov
特点:
最常用的蛋白质序列比对工具,支持搜索 nr(非冗余蛋白库)、Swiss-Prot 等数据库。
提供 E-value、Identity、保守域(CDD)注释。
适用场景:
快速查找同源蛋白,验证新蛋白功能。
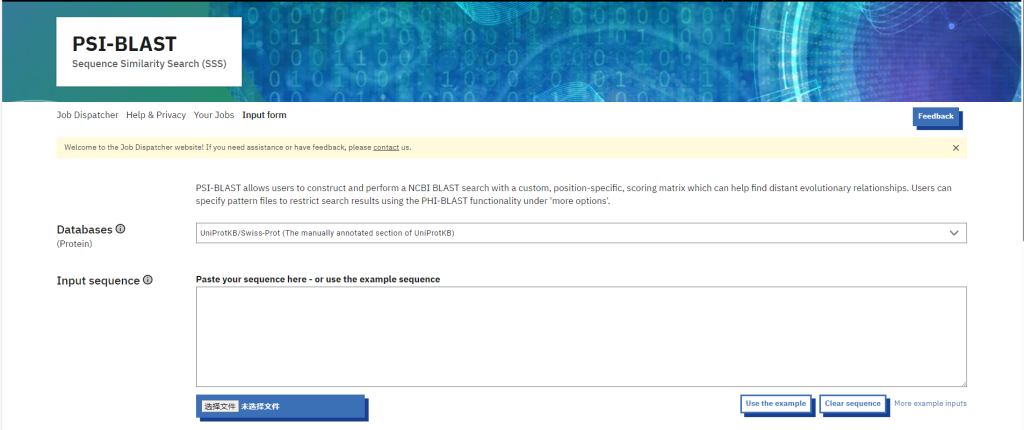
2 EBI PSI-BLAST
网址: https://www.ebi.ac.uk/jdispatcher/sss/psiblast
特点:
迭代搜索:通过多轮比对优化结果,每轮基于前一轮的PSSM进行新搜索。
检测远缘同源:比标准BLAST更适合发现进化距离较远的蛋白质。
自定义参数:可调整E值、迭代次数、数据库选择等。
适用场景:
适用于发现低相似性但功能相关的蛋白质序列
蛋白质的精确比对。
3. HHblits(基于HMM的超级加速版)
网址: https://toolkit.tuebingen.mpg.de/tools/hhblits
特点:
比PSI-BLAST更快更敏感,使用隐马尔可夫模型(HMM)。
适用场景:
宏基因组数据中的蛋白功能注释。
4. UniProt BLAST
网址: https://www.uniprot.org/blast/
特点:
专注于蛋白质序列比对,数据库涵盖Swiss-Prot、TrEMBL等高质量蛋白数据。
提供功能域注释(如PFAM、InterPro)。
适用场景:
蛋白质功能预测或结构域分析。
5. Clustal Omega
网址: https://www.ebi.ac.uk/Tools/msa/clustalo
特点:
经典工具,适合中等规模蛋白家族比对(<10万条序列)。
适用场景:
构建进化树前的序列对齐。
6. HMMER(蛋白家族搜索)
网址: https://www.ebi.ac.uk/Tools/hmmer/
特点:
基于隐马尔可夫模型(HMM),搜索Pfam等数据库。
适用场景:
鉴定蛋白家族或功能域(如激酶、GPCR)。
7.NCBI BLAST+
网址:https://www.ebi.ac.uk/jdispatcher/sss/ncbiblast
特点:
提供交互式结果可视化(如比对覆盖度、E 值分布等)。
适用场景:
适合中小规模数据分析。